Un nuovo modello cattura le molecole di glicano i cui movimenti proteggono gran parte della proteina spike dalle cellule della difesa immunitaria
La caratteristica principale di Sars-CoV-2 è la proteina spinale, che si estende su tutta la superficie e le consente di colpire e infettare le cellule umane. Molte ricerche hanno portato a modelli statici dettagliati della proteina spike, ma questi modelli non catturano l’elasticità della proteina spike stessa, né i movimenti delle catene glicaniche protettive delle molecole di zucchero che la incapsulano. Un nuovo modello dettagliato della superficie della proteina SARS-CoV-2, sviluppato dagli scienziati del Max Planck Institute for Biophysics di Francoforte sul Meno, mostra vulnerabilità precedentemente sconosciute che potrebbero portare allo sviluppo di un vaccino.
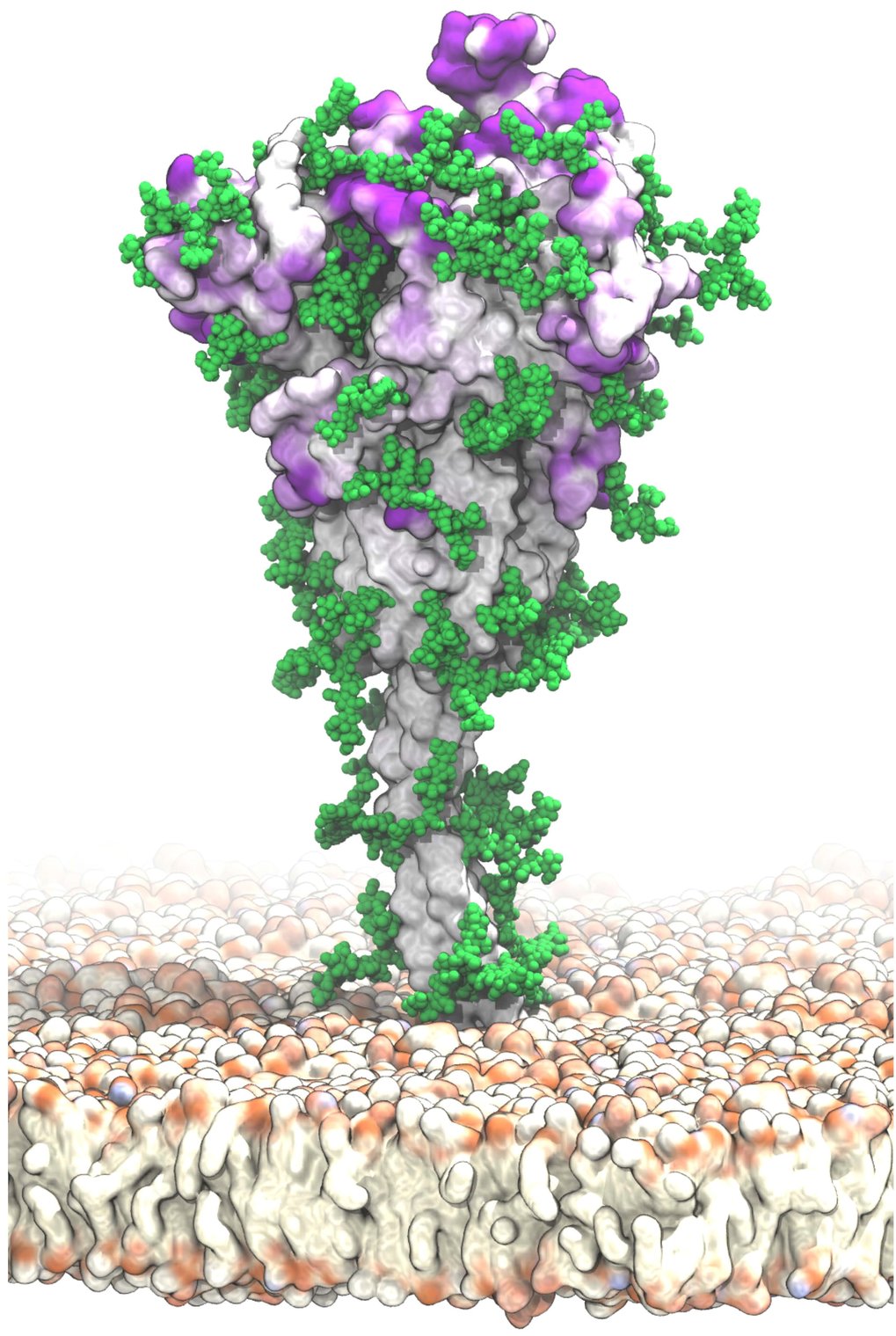
SARS-CoV-2 è associato alla membrana virale con un gambo sottile. Macchie viola intense sulla superficie indicano potenziali bersagli di anticorpi non protetti dalle catene glicaniche di molecole di zucchero mostrate in verde.
© MPI per Biophysics / Mateusz Sikora et al.
Per aiutare lo sviluppo del vaccino, Matthews Secura e colleghi hanno deciso di identificare potenziali nuovi siti bersaglio sulla superficie della proteina appuntita. Per fare ciò, hanno sviluppato simulazioni dinamiche molecolari che catturano l’intera struttura proteica e i movimenti dei picchi in un ambiente realistico.
Questa simulazione mostra che i glicani sulla proteina spike agiscono come uno scudo protettivo dinamico che aiuta il virus a eludere il sistema immunitario umano. Come con i tergicristalli per auto, i glicani coprono la superficie del picco quasi completamente sbattendo avanti e indietro, anche se ha poca copertura in ogni momento.
Combinando simulazioni dinamiche delle proteine spinali con ulteriori analisi bioinformatiche, i ricercatori hanno identificato i siti sulla superficie delle proteine spinali meno protetti dagli scudi glicani. Alcuni siti scoperti sono già stati identificati in ricerche precedenti, ma alcuni sono nuovi. La fattibilità di molti di questi nuovi siti è stata confermata da altri gruppi di ricerca in successivi esperimenti di laboratorio. “Siamo in una fase di una pandemia che cambia costantemente con l’emergere di nuovi tipi di SARS-CoV-2, con mutazioni che prendono di mira specificamente la proteina spike”, afferma Sikora. “Il nostro approccio può supportare la progettazione di vaccini e anticorpi terapeutici, soprattutto quando i metodi consolidati falliscono”. Il metodo sviluppato per questo studio può essere utilizzato anche per identificare potenziali debolezze in altre proteine virali.

“Specialista televisivo. Amichevole fanatico del web. Studioso di cibo. Drogato estremo di caffè.”





More Stories
Erasmus MC Sophia inizia con la diagnosi rapida di bambini molto malati
Come i defibrillatori leadless stanno cambiando la vita dei pazienti in tutto il mondo
Mucche francesi infette dall’antrace